
近年来,随着多种测序技术的结合和分析策略的进步,越来越多的物种完成了高质量基因组参考序列的组装。植物中许多物种已经从“一个物种,一个基因组”的时代进入了“一个物种,多个基因组”的“泛基因组时代”(pan-genomic era)。特别是如小麦、玉米和水稻等主要作物,在近缘种、亚种、个体水平都已经完成多个参考基因组的组装和基因的注释。在泛基因组时代,如何整合诸多的参考基因组信息并将这些宝贵的数据利用起来,是我们目前面临的新挑战。
近日,我院小麦研究中心郭伟龙副教授作为通讯作者在Molecular Plant杂志在线发表了题为“A Collinearity-incorporating Homology Inference Strategy for Connecting Emerging Assemblies in Triticeae Tribe as a Pilot Practice in the Plant Pangenomic Era”的研究论文。

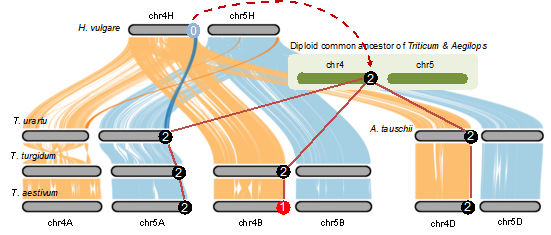
该研究提出了整合共线性的同源基因推断新策略(GeneTribe)并构建了小麦族同源基因数据库(Triticeae-GeneTribe)。基于小麦族物种基因组间和亚基因组间的共线性分析,该研究提出了六倍体普通小麦的“4A-5A-7B染色体重排”是两次染色体易位事件的结果,并明确了重排的基因组区间的精细边界;同时研究了春化基因Vrn2的复杂进化历史,提出Vrn2同源基因在普通小麦基因组中的复杂分布是包含串联重复、多倍化、染色体易位和基因丢失在内的一系列事件叠加的结果。该工作为泛基因组时代的植物比较基因组学研究和功能基因挖掘提供了新思路。
我院郭伟龙副教授为该论文的通讯作者,博士研究生陈永明为第一作者。小麦研究中心研究生宋皖君、谢小明(参与时为本科生)、王梓豪和博士后关攀峰为共同作者。我院孙其信教授、倪中福教授、彭惠茹教授和中国科学院植物研究所焦远年研究员对该工作进行了指导和帮助。该工作得到了国家自然科学基金(31991210和31701415)项目的资助。
原文链接:https://doi.org/10.1016/j.molp.2020.09.019